
Science with Passion
App-Nr.: VBS0083
Version 1 04/2024
Qualitätskontrolle von Oligonukleotiden mittels HPLC gekoppelt mit UV- und MS-Detektion
J. Kramer1, U. Krop1, G. Greco1, T. Pöhlmann2, C. Matthäus2; kramer@knauer.net
1 KNAUER Wissenschaftliche Geräte GmbH, Hegauer Weg 38, 14163 Berlin
2 BianoGMP GmbH, Ronneburger Straße 74, 07546 Gera, Germany

Zusammenfassung
Die Quantifizierung von Oligonukleotiden kann leicht durch UV-Detektion erfolgen, da Oligonukleotide eine starke Absorption bei 260 nm zeigen. Die Erweiterung der UV-Methode zur Qualitätskontrolle mit Massenspektrometrie führt zu einer Massenbestätigung und Identifizierung von Verunreinigungen.
Einführung
In den letzten zwei Jahrzehnten sind Oligonukleotide zu einem wichtigen Werkzeug in der Grundlagenforschung und einer leistungsstarken Technologie für die Arzneimittelentwicklung geworden. Diese auf Oligonukleotiden basierenden Medikamente haben ihre therapeutische Wirksamkeit bewiesen und ein ausgezeichnetes Toxikologieprofil gezeigt. Ein Aspekt des Herstellungsprozesses ist die Reinigung des aktiven pharmazeutischen Wirkstoffs (API) und seine Trennung von unerwünschten Nebenprodukten. Um die erfolgreiche Synthese und Reinigung zu gewährleisten, ist eine analytische Qualitätskontrolle zwingend erforderlich. Die Qualitätskontrolle erfordert eine zuverlässige Bestätigung der Oligonukleotidmasse sowie die Quantifizierung von Verunreinigungen. Abgebrochene Sequenzen, insbesondere N-1, sind oft nicht basenliniengetrennt, was eine zuverlässige Quantifizierung erschwert. Andere Verunreinigungen, die aufgrund von Abbauprozessen auftreten, wie zum Beispiel Depurinierung, können nicht ausreichend von dem Hauptpeak getrennt werden. Solche koelutierenden Verunreinigungen können genau mit einem Massenspektrometer quantifiziert werden. Die Flüssigkeitschromatographie, insbesondere die Ion-Paar-reverse Phase (IP-RP) HPLC, ist eine der am häufigsten verwendeten Methoden zur Analyse von Oligonukleotidverunreinigungen [1]. Die nachfolgende Anwendung zeigt einen möglichen Arbeitsablauf, der ein beispielhaftes Sample verwendet, zur Charakterisierung von Oligonukleotiden und zur Quantifizierung von Verunreinigungen über LC-UV und LC-MS. In dieser Arbeit haben wir mit der BianoGMP GmbH zusammengearbeitet. Das Unternehmen ist auf die Produktion von hochreinen und qualitativ hochwertigen Oligonukleotiden spezialisiert und verfügt über viele Jahre Erfahrung in der Entwicklung therapeutischer Oligonukleotide mit einem Fokus auf GMP-Dienstleistungen und analytische Methoden für Oligonukleotide.
Probenvorbereitung
Die verwendete Probe war ein 26-mer Oligonukleotid mit einer nominalen Masse von 8369,9 Da, bereitgestellt von BianoGMP GmbH in Gera, Deutschland. Eine Lösung von 10 mg/ml wurde hergestellt, indem 2,5 ml Wasser zu 25 mg Referenzstandard hinzugefügt wurden. Anschließend wurde die 10 mg/ml Lösung im Verhältnis 1:10 mit Wasser verdünnt, um eine Konzentration von 1 mg/ml zu erreichen. Die 1:10 Verdünnung enthielt 119,5 nmol Substrat in 1 ml. Diese wurde auf 25 nmol/ml verdünnt. Daher wurden 209,2 μl der 1 mg/ml Lösung auf 1 ml aufgefüllt. Für die Analyse wurden 2 μl der 25 nmol/ml Lösung injiziert, was einer absoluten Substanzmenge von 50 pmol entspricht.
Ergebnisse
Das Oligonukleotid wurde unter Bedingungen der Ionenaustausch-Reversed-Phase-Analyse mit einer wässrigen Lösung, die Hexafluor-Isopropanol (HFIP) und Triethylamin (TEA) als Eluent A und 100 % Methanol als Eluent B enthält, analysiert. Ein Injektionsvolumen von 2 µl der Referenzlösung (25 nmol/ml) wurde injiziert. Die UV-Absorption bei 260 nm wurde aufgezeichnet. Abb. 1 zeigt das UV-Chromatogramm der injizierten Probe. Peakfläche, Peakhöhe und Retentionszeit können von der Software berechnet und zur Quantifizierung verwendet werden.
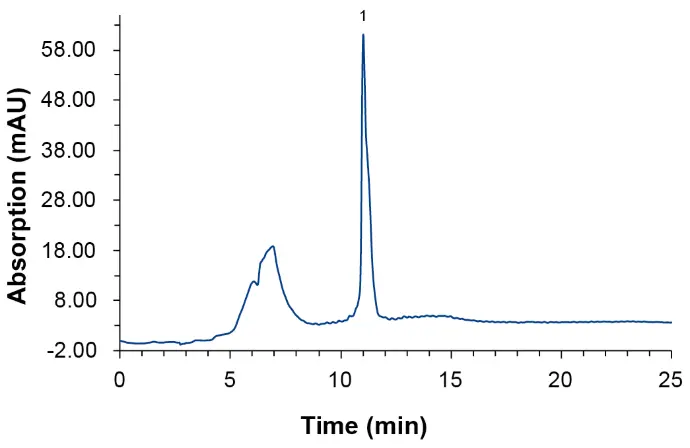
Abb. 1 UV-Trace der Oligonukleotidprobe bei 260 nm
Nach der UV-Detektion wurde die Probe weiter mit einem Triple-Quadrupol-Massenspektrometer analysiert. Die Probe wurde mit Elektrospray-Ionisation (ESI) mit negativer Polarität analysiert. Abb. 2 zeigt den Gesamt-Ionenstrom (TIC) für die Oligonukleotidprobe.
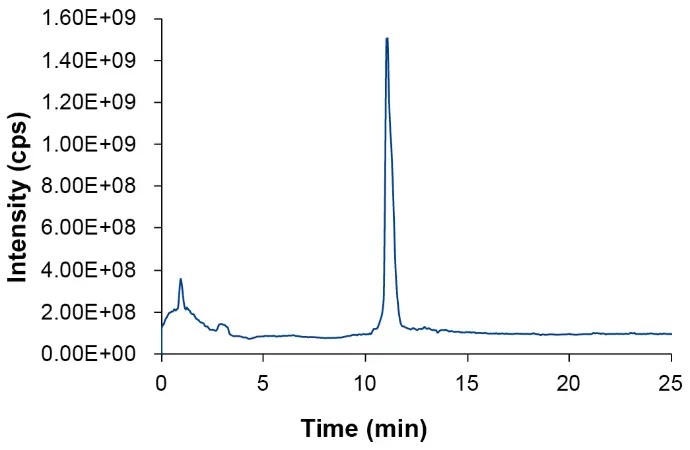
Abb. 2 TIC der Oligonukleotidprobe
Der erste Quadrupol (Q1) des MS wurde für einen Scan im Bereich von 500 Da bis 1250 Da verwendet. Das Spektrum (Abb. 3) wurde basierend auf der Retentionszeit, wie im TIC zu sehen, extrahiert. Das Massenspektrum zeigte deutlich die Reihe der deprotonierten Ladungszustände (z = 7 bis 15). Die Verteilung der Ladungszustände kann je nach dem spezifischen untersuchten Oligonukleotid und anderen Parametern wie dem pH-Wert der Lösung schwanken.
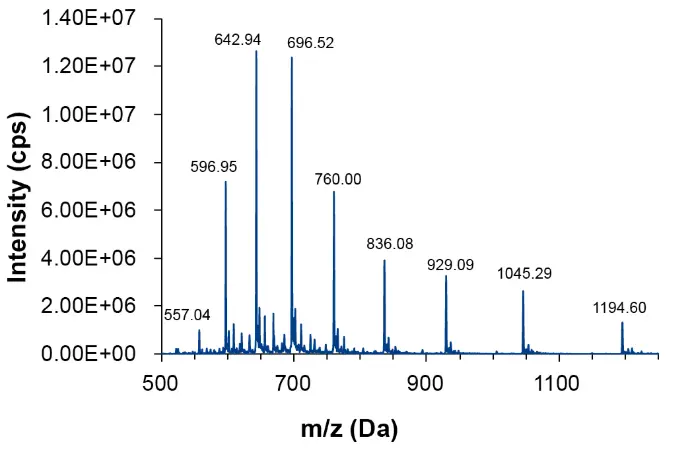
Abb. 3 Q1-Spektrum der Oligonukleotidprobe von 10,95 bis 11,27 Minuten, m/z 500–1250 Da
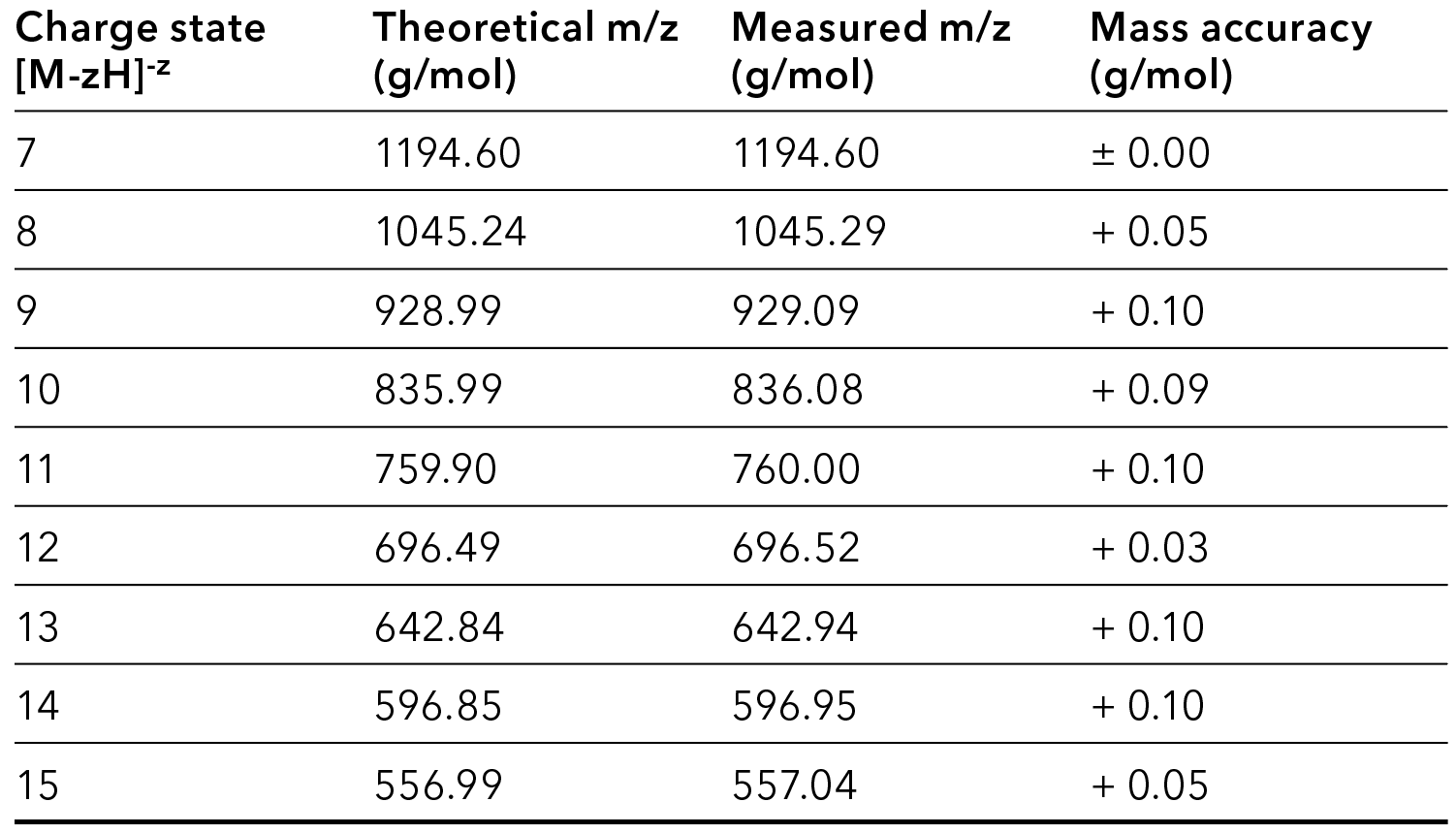
Der Vergleich von theoretischer und gemessener Masse wurde zur Bestätigung der Oligonukleotidmasse verwendet. Die Abweichungen waren genau für die Bestätigung des Analyten durch die Detektion von m/z-Spezies.
Tab. 1 Vergleich der berechneten und bestimmten m/z-Werte für das Hauptprodukt, Q1-Scan
Abgebrochene Sequenzen (N-x) sind oft chromatographisch nicht gut von der vollständigen Sequenz (N) oder voneinander getrennt, aufgrund ihrer geringen Massendifferenzen. Dies gilt insbesondere für N-1, wo kleine Mengen durch die vordere Flanke des Hauptproduktgipfels mit viel größerer Peakfläche verdeckt werden. Die einzelnen Oligonukleotidverunreinigungen N-1 für die Ladungszustände mit der höchsten Intensität, hier m/z=12 und m/z=13, wurden untersucht. Das detaillierte Spektrum für N-1 bei einem Ladungszustand von m/z=13 im Vergleich zum Hauptpeak ist in Abb. 4 dargestellt. Die Massengenauigkeit der theoretischen und gemessenen Masse beträgt 0,10 Da für den Hauptproduktpeak und 0,01 Da für den Peak der N-1-Verunreinigung.
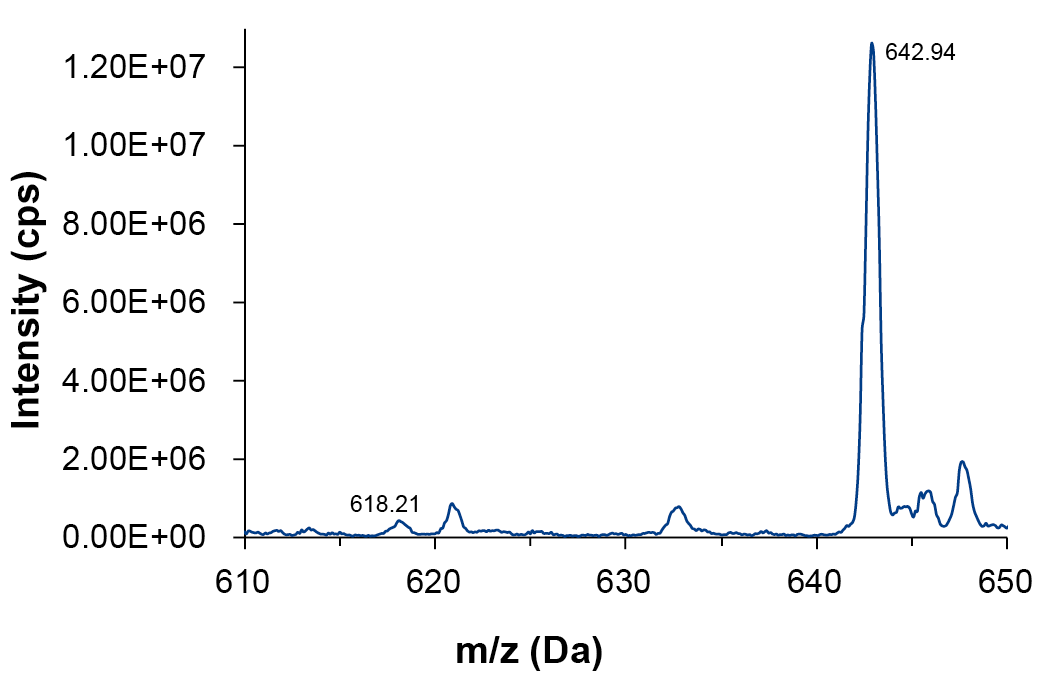
Abb. 4 Q1-Spektrum in einem Bereich von 610 – 650 Da, m/z=13, Hauptpeak (642,94) und N-1-Verunreinigung (618,21)
Das verwendete Oligonukleotid ist ein voll-thioatiertes 26-Mer. Daher müssen depurinierte (NDEP) Abbauprodukte und die Rücksubstitution von Schwefel mit Sauerstoff (NPO) berücksichtigt werden. Der intensivste Ladungszustand, der im Q1-Scan sichtbar ist, hier bei 642,94 Da (z=13), wurde für die Quantifizierung dieser koeluierten Verunreinigungen ausgewählt. Die Quantifizierung wurde unter Verwendung einer multiplen Ionenüberwachung (Q1MI) durchgeführt. Die extrahierten Einzelionen, NDEP und NPO, sowie der Hauptpeak bei m/z=13 aus dem Q1MI-Experiment sind in Abb. 5 dargestellt.
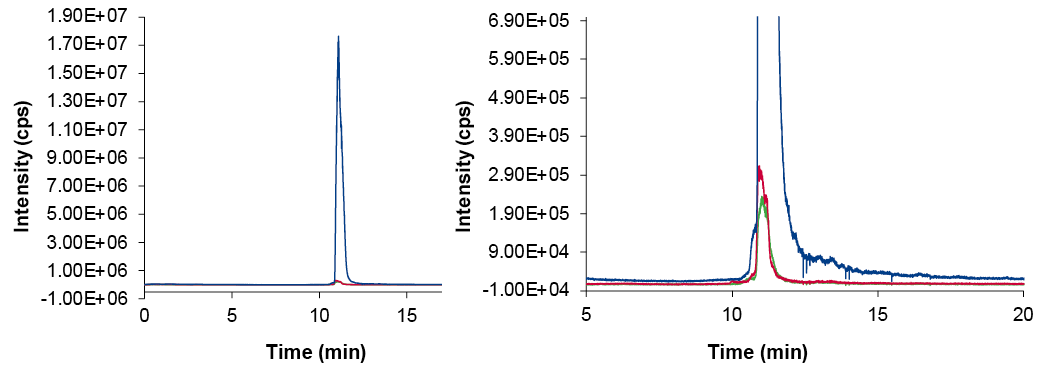
Abb. 5 XIC von SIM bei m/z=13 (links), vergrößerte Ansicht der XIC (rechts), blau – Hauptpeak, rot – NPO, grün – NDEP
Die Menge der Verunreinigung wurde als Prozentsatz in Bezug auf die Fläche des Hauptgipfels (Abb. 6) berechnet, aber natürlich kann auch eine gemeinsame Kalibrierung auf verschiedenen Konzentrationsniveaus verwendet werden.
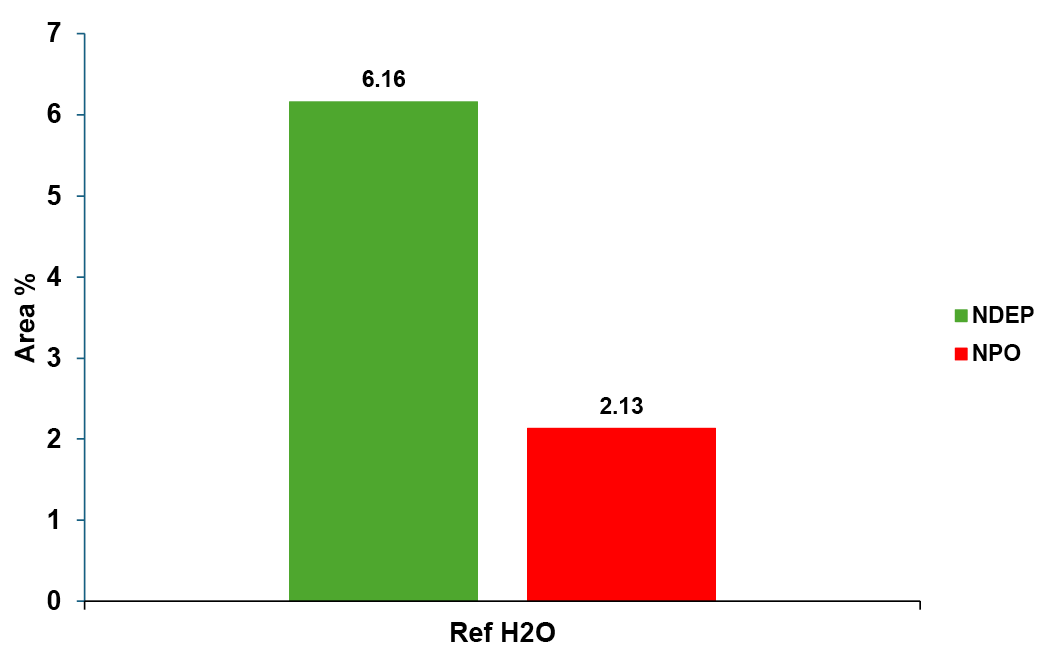
Abb. 6 Flächenanteil % der Verunreinigungen aus SIM bei m/z=13 im Verhältnis zum Hauptpeak (100%)
In der Referenzlösung wurden Werte für NDEP von etwa 6 % im Verhältnis zum Hauptpeak und für NPO von etwa 2 % im Verhältnis zum Hauptpeak erwartet.
Fazit
Erweiterung der HPLC-UV-Methode zur Qualitätskontrolle mit MS-Vorteilen bei der Massenbestätigung und Identifizierung von Verunreinigungen. Verunreinigungen wie abgebrochene Sequenzen (N-1), Depurinierungsprodukte oder andere Modifikationen können untersucht werden, obwohl sie nicht oder nicht ausreichend von der vollständigen Sequenz (N) getrennt sind. HPLC in Kombination mit negativer ESI-MS bietet eine zuverlässige Methode mit einzigartigen Möglichkeiten zur Trennung und Identifizierung von (therapeutischen) Oligonukleotiden. Der beschriebene Arbeitsablauf nutzte nur das erste Quadrupol, aber MS/MS- oder MRM-Experimente können nützlich sein für die quantitative Analyse von Oligonukleotidverunreinigungen in niedrigen Konzentrationen und koeluierten Substanzen.
Material und Methoden
Tab. 2 Systemkonfiguration
Instrument | Beschreibung | Artikel-Nr. |
Pumpe
| AZURA® P 8.1L UHPLC |
|
Autosampler | AZURA® AS 6.1L, 1240 bar, cooling/heating version
|
|
UV-Detektor | AZURA® DAD 6.1L
|
|
UV-Flusszelle
| Standard KNAUER LightGuide UV Flusszellenpatrone
|
|
Thermostat
| AZURA® CT2.1 |
|
LC software
| ClarityChrom 9.0.0 - Workstation, Steuerung des Autosamplers enthalten
|
|
MS
| Sciex Triple Quad™ 5500+ System – QTRAP® Ready
| – |
MS Software
| Sciex OS 3.2
| – |
Spalte
| Waters XBridge Oligonukleotid BEH C18 Säule, 50 x 4,6 mm ID, 2,5 µm, 130 Å
| – |

Abb. 6 FPLC-Systemkonfiguration
Tab. 1 FPLC-Methodeneinstellungen
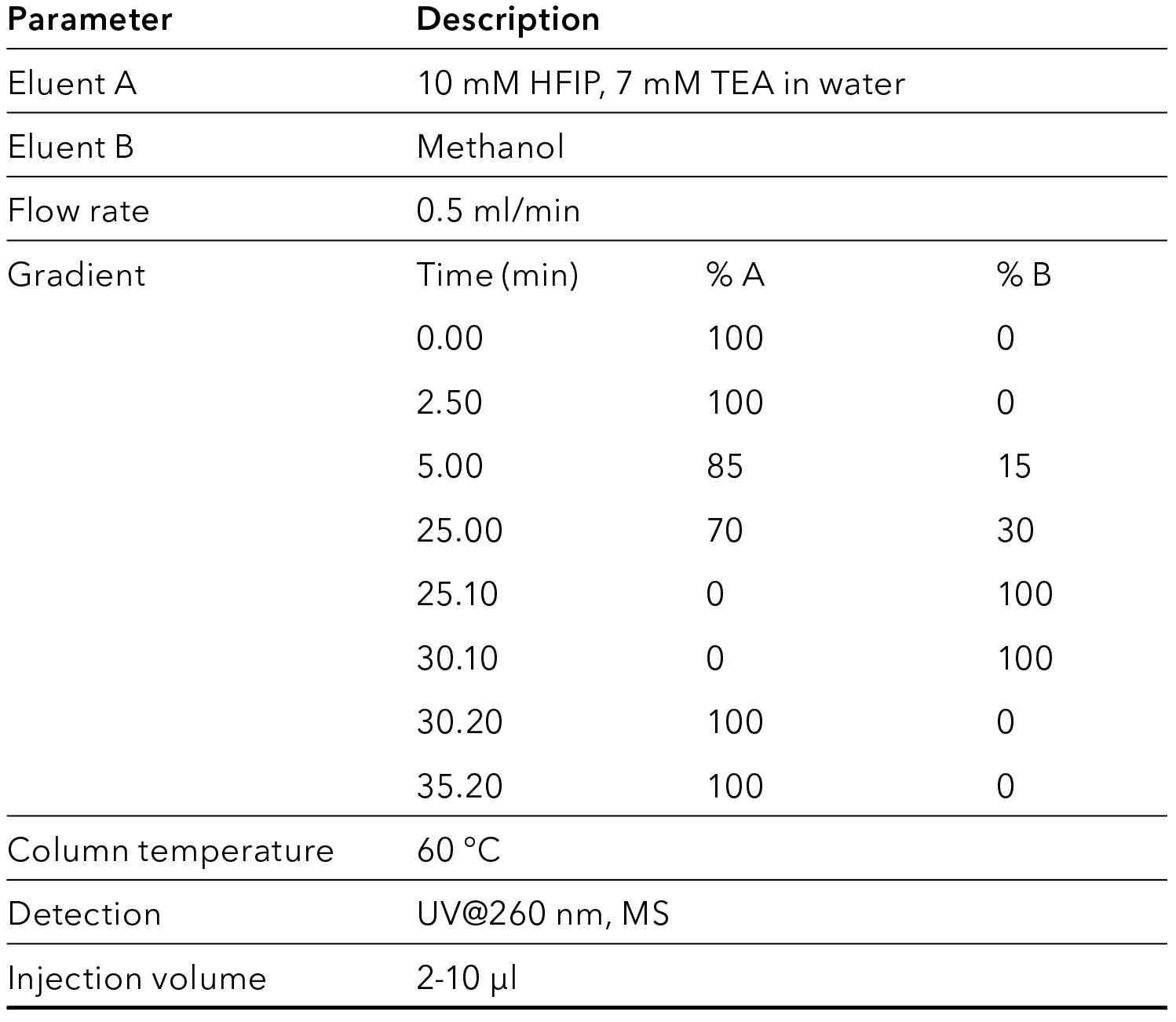
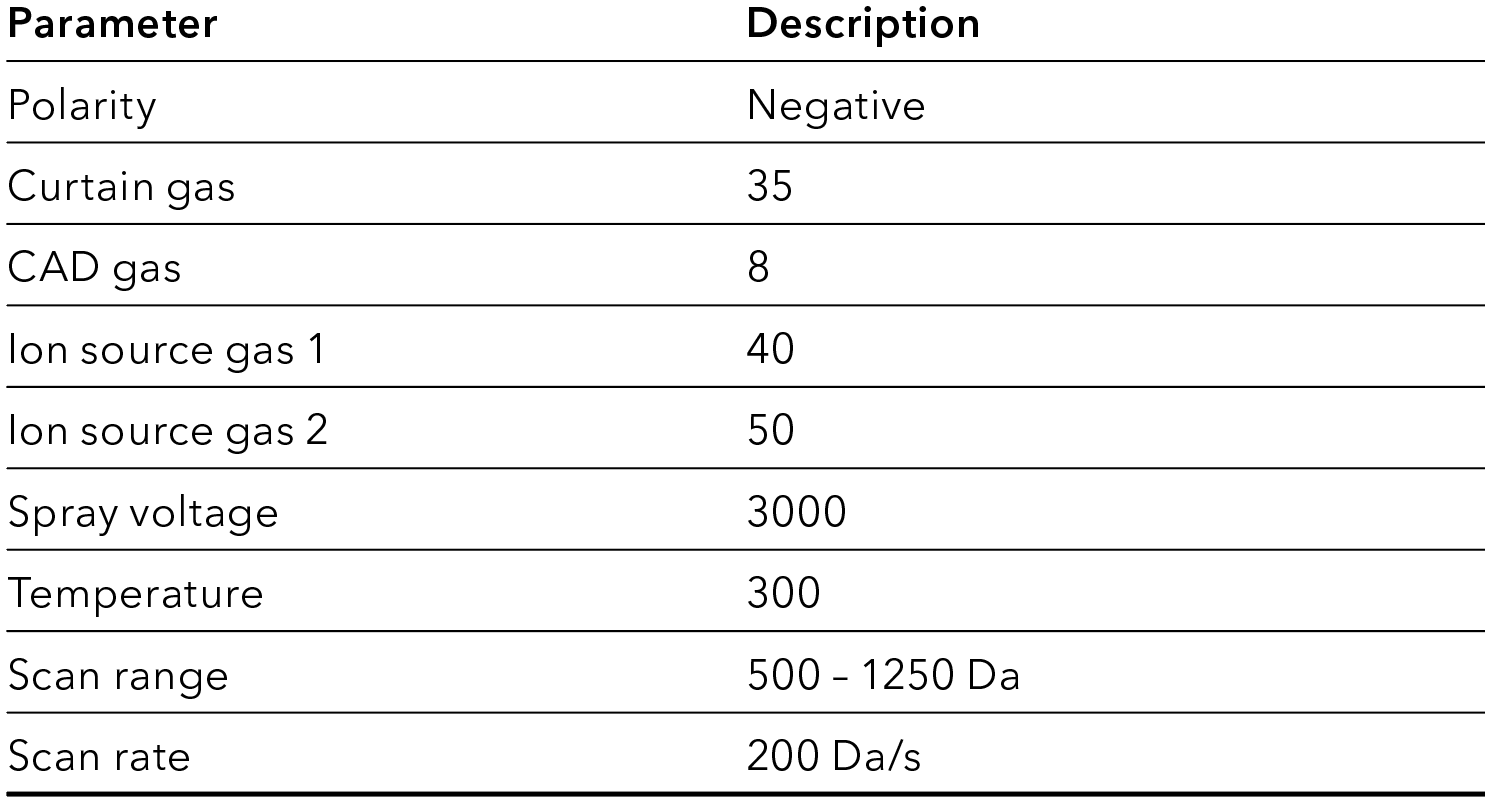
Tab. 4 MS-Methodeneinstellungen
Referenzen
[1] Journal of Chromatography B 1169 (2021) 122611; Bestimmung einzelner Oligonukleotidverunreinigungen durch kleine Ammonium-Ionenpaar-RP HPLC MS und MS/MS: n - 1 Verunreinigungen; Stilianos G. Roussis, Andrew A. Rodriguez, Claus Rentel; https://doi.org/10.1016/j.jchromb.2021.122611
Anwendungsdetails
Methode | LC-MS |
Modus | RP |
Substanzen | Oligonukleotid |
CAS-Nummer | n/a |



